Non sarà divertente come un’intelligenza artificiale che crea immagini di Gesù-Gamberetto, non ci farà sentire scaltri quanto un chatbot che scrive noiose email di lavoro al posto nostro, ma dovremmo prestare un po’ più di attenzione a quello che sta succedendo nella bioinformatica, in pratica quando l’intelligenza artificiale viene usata in biologia.
Un argomento poco sexy perché è più difficile da capire e perché sembra di interesse esclusivo della comunità scientifica dedita alla produzione di queste bellissime ma incomprensibili immagini 3D della struttura delle proteine. Ma ve lo rendiamo sexy noi, spiegandovi meglio di cosa si tratta.
Partiamo dalla notizia del momento: l’8 maggio scorso DeepMind, la divisione di Alphabet/Google dedicata all’intelligenza artificiale, presenta AlphaFold 3 (AF3, per gli amici), con tanto di pubblicazione su Nature, descrivendolo come “a revolutionary model that can predict the structure and interactions of all life’s molecules with unprecedented accuracy”.
Google e Demis Hassabis, chief executive di DeepMind, incalzano, spiegando che cosa si in intende con quell’ “all life’s molecules”: se le versioni precedenti di AlphaFold erano in grado di predire “solo” la struttura delle proteine, AlphaFold 3 può modellizzare DNA, RNA e molecole più piccole chiamate ligandi, ampliando le potenzialità del modello per uso scientifico.
La comunità scientifica esulta (“grandi avanzamenti per la ricerca scientifica in vista”), dibatte (“eh però, non pubblicano il codice”), si arrovella (“aaaah qualcuno pensi alle armi biologiche!”).
Ma che vuol dire? Che cosa faceva AlphaFold2? A cosa serve sapere la struttura delle proteine?
Il problema del folding delle proteine
Le proteine, ingrediente indispensabile per l’esistenza degli esseri viventi, sono macromolecole costituite da catene di amminoacidi. All’interno degli organismi, svolgono una vasta gamma di funzioni: da quella plastica e strutturale, alla catalisi di reazioni chimiche all’interno delle cellule, alla generazione e modulazione delle risposte agli stimoli interni ed esterni, al trasporto di molecole da un luogo all’altro della cellula. La funzione che svolge una proteina, sia essa un enzima, un trasportatore, un recettore o una proteina strutturale, è strettamente legata alla sua struttura tridimensionale, tanto che in biologia molecolare si dice che “la struttura di una proteina è essa stessa funzione”.
Come ha spiegato la logonner Chiara Di Lucente su Wired:
“sebbene le singole unità di cui sono composte – gli aminoacidi – siano solo 20, disposti in sequenze precise, in natura esistono milioni di proteine diverse, ognuna delle quali ha caratteristiche e funzioni differenti. Questo è possibile perché gli amminoacidi si arrangiano nello spazio in maniera diversa, a seconda delle loro caratteristiche chimiche: pertanto, a ogni sequenza di amminoacidi di cui è costituita una proteina, corrisponderà una struttura diversa. Comprendere la configurazione tridimensionale di una proteina è fondamentale nella ricerca, perché essa fornisce informazioni sulla sua funzione e su come modificarla, bloccarla o regolarla.”
Anche per sviluppare nuovi farmaci dobbiamo conoscere la struttura 3D delle proteine: la maggior parte dei farmaci, infatti, sono piccole molecole note come ligandi che si legano alle proteine per modificare il modo in cui esse funzionano in processi patologici. Un concetto molto noto in biologia è quello del cosiddetto modello “lock-and-key”, secondo cui una proteina, per funzionare correttamente con un certo ligando, deve interagire con esso a livello strutturale, nello stesso modo in cui una chiave entra nella serratura. Trovare un farmaco o un vaccino che abbia un certo effetto su una proteina, quindi, è un po’ come trovare la giusta chiave per una serratura da aprire, o viceversa da chiudere o bloccare. Ipotizziamo una proteina essenziale per la sopravvivenza di un virus (ad esempio, una proteina che sintetizza il suo DNA), che avrà un certo tipo di “serratura”. Trovando la giusta chiave per quella serratura, sarà possibile aprirla, oppure bloccarla. Trovando la chiave in grado di bloccare la serratura, questa sarà sempre chiusa e, tornando all’esempio della nostra proteina virale, la replicazione del virus sarà inibita e l’infezione scongiurata. Tutto questo, ovviamente, se si conoscono approfonditamente la forma e la struttura della nostra serratura-proteina. Non è affatto un caso che AlphaFold abbia aiutato già la ricerca a contrastare i virus, compreso l’attuale coronavirus protagonista della pandemia da Covid-19.
 La capacità di predire quale struttura tridimensionale assumerà la proteina sulla base della sequenza di aminoacidi da cui è composta, secondo il processo del cosiddetto folding proteico, richiedeva l’applicazione di metodi sperimentali complessi, lunghi e costosi. D’altro canto, l’approccio computazionale forniva solo soluzioni limitate, fino all’avvento di AlphaFold 2. Grazie al machine learning, infatti, AlphaFold 2 è in grado di integrare le conoscenze fisiche e biologiche sulla struttura delle proteine (e quindi, per esempio, il modo in cui statisticamente una data sequenza di amminoacidi si dispone nello spazio), con simulazioni e confronti tra molteplici sequenze di amminoacidi, in modo da generare un algoritmo di deep learning in grado da predire con più precisione il folding delle sequenze amminoacidiche.
La capacità di predire quale struttura tridimensionale assumerà la proteina sulla base della sequenza di aminoacidi da cui è composta, secondo il processo del cosiddetto folding proteico, richiedeva l’applicazione di metodi sperimentali complessi, lunghi e costosi. D’altro canto, l’approccio computazionale forniva solo soluzioni limitate, fino all’avvento di AlphaFold 2. Grazie al machine learning, infatti, AlphaFold 2 è in grado di integrare le conoscenze fisiche e biologiche sulla struttura delle proteine (e quindi, per esempio, il modo in cui statisticamente una data sequenza di amminoacidi si dispone nello spazio), con simulazioni e confronti tra molteplici sequenze di amminoacidi, in modo da generare un algoritmo di deep learning in grado da predire con più precisione il folding delle sequenze amminoacidiche.
In questo modo, il database (gratuito) di AlphaFold garantisce l’accesso a oltre 200 milioni di strutture proteiche, velocizzando il lavoro degli scienziati in numerosissimi ambiti della ricerca. In effetti abbiamo parlato di un’applicazione di campo medico, ma in realtà, il raggio di azione dello studio delle strutture proteiche è molto ampio, proprio perché esse sono una componente fondamentale degli esseri viventi: altri applicazioni potrebbero essere nell’agricoltura e nelle scienze dei materiali.
Le novità di AlphaFold 3
AlphaFold3 usa un nuovo tipo di machine learning chiamato modello di diffusione, lo stesso utilizzato nella generazione di immagini o di video da intelligenze artificiali più famose al grande pubblico come Stable Diffusion, Midjourney o Sora.
In pratica il modello è stato addestrato per apprendere un processo di riduzione del rumore dell’immagine. Come dati di addestramento ha ricevuto immagini a cui veniva aggiunto rumore (rumore gaussiano) e ha imparato a recuperare l’immagine invertendo il processo. Una volta imparato questo processo di de-noising lo può applicare alla generazione di nuove immagini. Parte generando un’immagine semi-casuale rumorosa da cui riesce a far emergere una previsione accurata dell’immagine più verosimile.
Stiamo parlando di immagini 3D di strutture molecolari, quindi, in pratica mentre in Midjourney inseriamo un prompt di testo per ottenere un’immagine, i ricercatori inseriscono un elenco di molecole che vogliono combinare e AlphaFold 3 genera un modello 3D della nuova struttura.
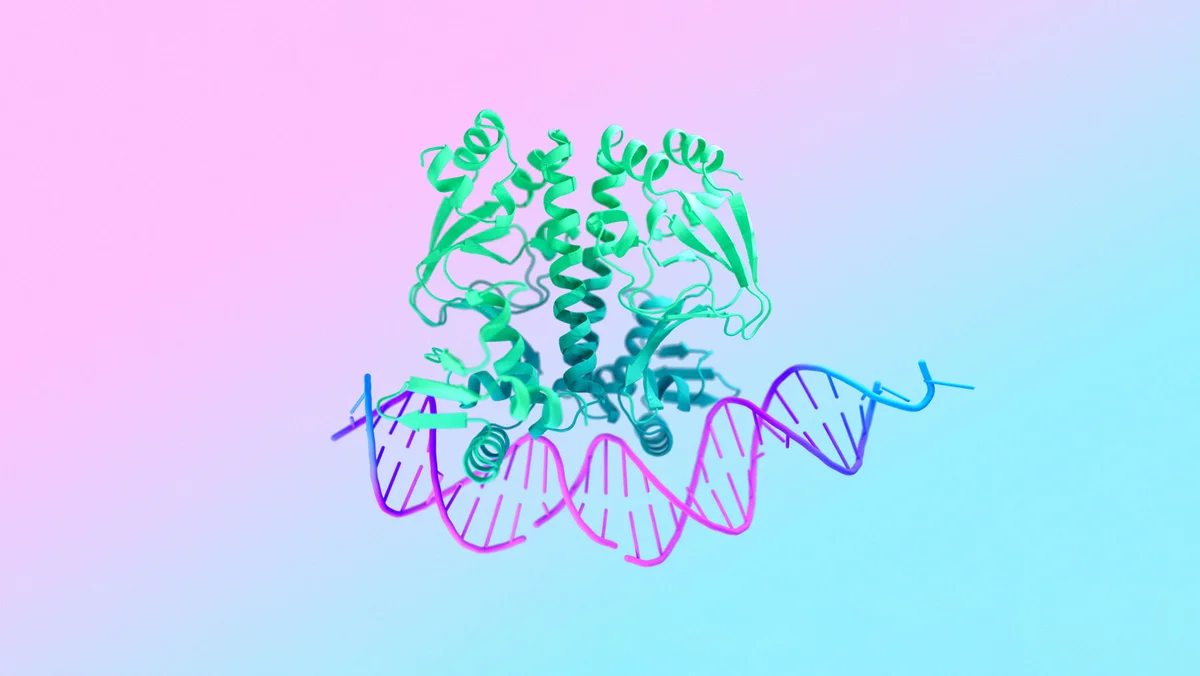
La grande novità dell’utilizzo del modello di diffusione è innanzitutto che risulta particolarmente efficace nelle predizioni (con un miglioramento del 50% nell’accuratezza di predizione). Grazie alla capacità di gestire un maggior quantitativo di input, genera predizioni a livello delle strutture atomiche di diverse molecole e macromolecole (come DNA, RNA, ligandi, ioni metallici e così via) e non solo le sequenze di amminoacidi. Ed è questa capacità che permette a AF3, quindi, di predire non solo la struttura di singole proteine, ma di altre molecole biologiche e soprattutto l’interazione tra di esse.
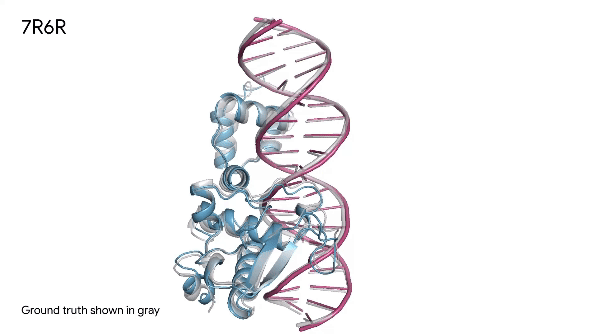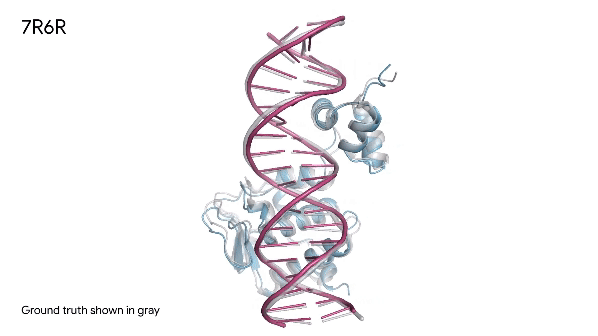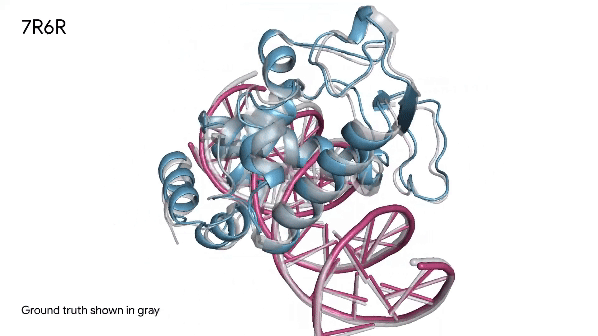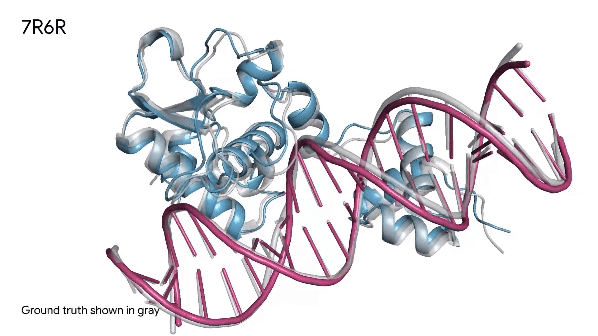
Le proprietà e le funzioni delle molecole nei sistemi biologici sono tipicamente il risultato di come interagiscono con altre molecole. Le proteine interagiscono con altre proteine, con DNA/RNA, molecole, ioni ecc, che a loro volta interagiscono tra loro e con altre molecole ancora. I farmaci spesso agiscono non sulle singole proteine ma proprio su queste complesse interazioni biomolecolari. Per comprenderle, la ricerca sperimentale può richiedere anni, ma se possiamo avere una simulazione realistica, sufficientemente accurata, ecco che la ricerca può spingere sull’acceleratore.
In sostanza, se sappiamo che una proteina esegue una certa funzione – magari legata a una malattia – grazie all’interazione con un ligando (il meccanismo lock-and-key di prima), lo studio di questa interazione a livello strutturale ci potrebbe dire cose molto interessanti sulla funzione della proteina: per esempio dove entra la chiave, che forma deve avere e la forma di altre chiavi in grado di bloccare la serratura. Tutto questo rappresenta una grande risorsa per il mondo della ricerca.
AlphaFold 2 era stato reso disponibile da DeepMind gratuitamente ai ricercatori, non è lo stesso per AF3 che è disponibile gratuitamente solo per uso non-commerciale e con un accesso limitato: massimo 10 predizioni biomolecolari al giorno, non collegate allo sviluppo di farmaci. E questo ha generato qualche borbottio…
Dal 2021 infatti la famiglia Alphabet comprende anche Isomorphic Labs, un’azienda che si occupa di drug discovery fondata dal CEO di Google DeepMind Hassabis. Isomorphic Labs utilizzerà AlphaFold 3 per progetti interni e in collaborazione con aziende farmaceutiche.
Un altro dei temi di dibattito è quello dei rischi per la biosicurezza, riconosciuti anche da Google secondo cui i modelli di AI “possano abbassare la barriera per gli attori di minacce e consentire loro, di concerto con altre tecnologie, di progettare e ingegnerizzare agenti patogeni e tossine più trasmissibili o dannosi”.
Google afferma che sta collaborando con la comunità scientifica e i leader politici per utilizzare il modello in modo responsabile.
Una considerazione personale sull’impatto mediatico di queste innovazioni: ci lasciamo incantare dalla magia di un’intelligenza artificiale come ChatGPT che riesce a sembrare umana grazie alle sue capacità linguistiche, riesce a imitare alcune delle capacità dell’intelligenza umana e possiamo interagire con “lei” in maniera intuitiva, proprio come se fosse una persona. Ma al di là dell’incanto non è ancora del tutto chiaro quali possono essere l’utilità, il modello di business e la sostenibilità di ChatGPT (o di un’AI text-to-image).
In questo caso invece abbiamo un’intelligenza artificiale che diventa uno strumento con una finalità chiara e che ci permette di fare qualcosa che con la sola intelligenza umana (e i limiti di risorse e di tempo) non potremmo fare. Ma la notizia rimane in una bolla isolata.
Voi ne avevate sentito parlare?
Per approfondire
- Abramson, J. et al. Nature https://doi.org/10.1038/s41586-024-07487-w (2024).
- Jumper, J. et al. Nature 596, 583–589 (2021).
- https://www.nature.com/articles/d41586-024-01383-z
- https://www.theverge.com/2024/5/8/24152088/google-deepmind-ai-model-predict-molecular-structure-alphafold
- https://alphafold.ebi.ac.uk/
- https://blog.google/technology/ai/google-deepmind-isomorphic-alphafold-3-ai-model/
- https://www.wired.it/article/intelligenza-artificiale-di-deepmind-puo-prevedere-la-struttura-di-quasi-tutte-le-proteine-note/
- https://time.com/6975934/google-deepmind-alphafold-3-ai/